Biologie computationnelle
Le groupe de biologie computationnelle aborde les problèmes biologiques à l'aide de méthodes computationnelles, en se concentrant sur les mitochondries en tant qu'organites métaboliques importants et sur l'évolution des systèmes biologiques.
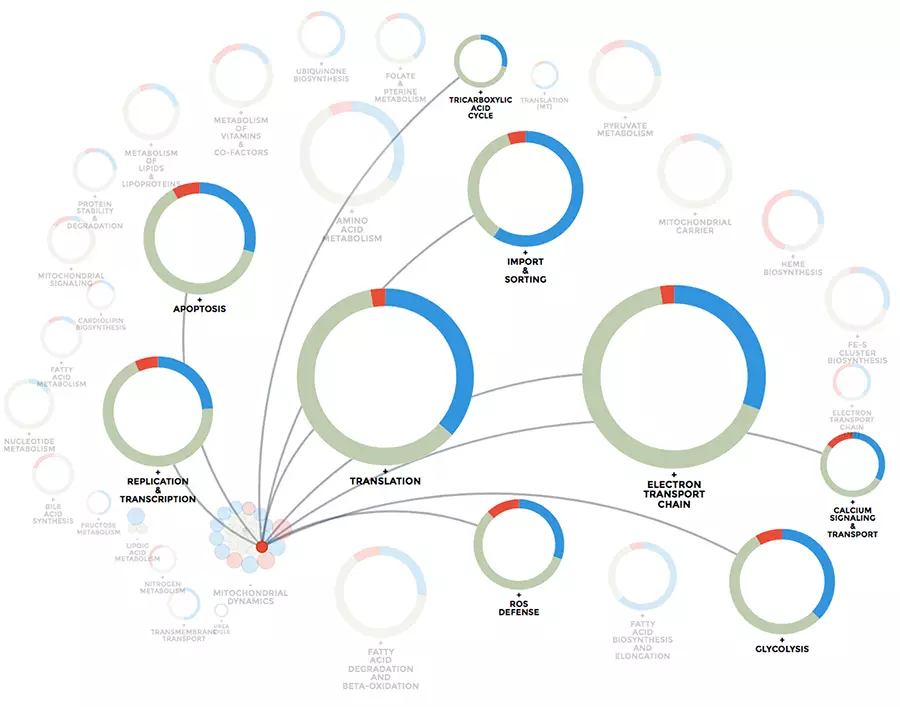
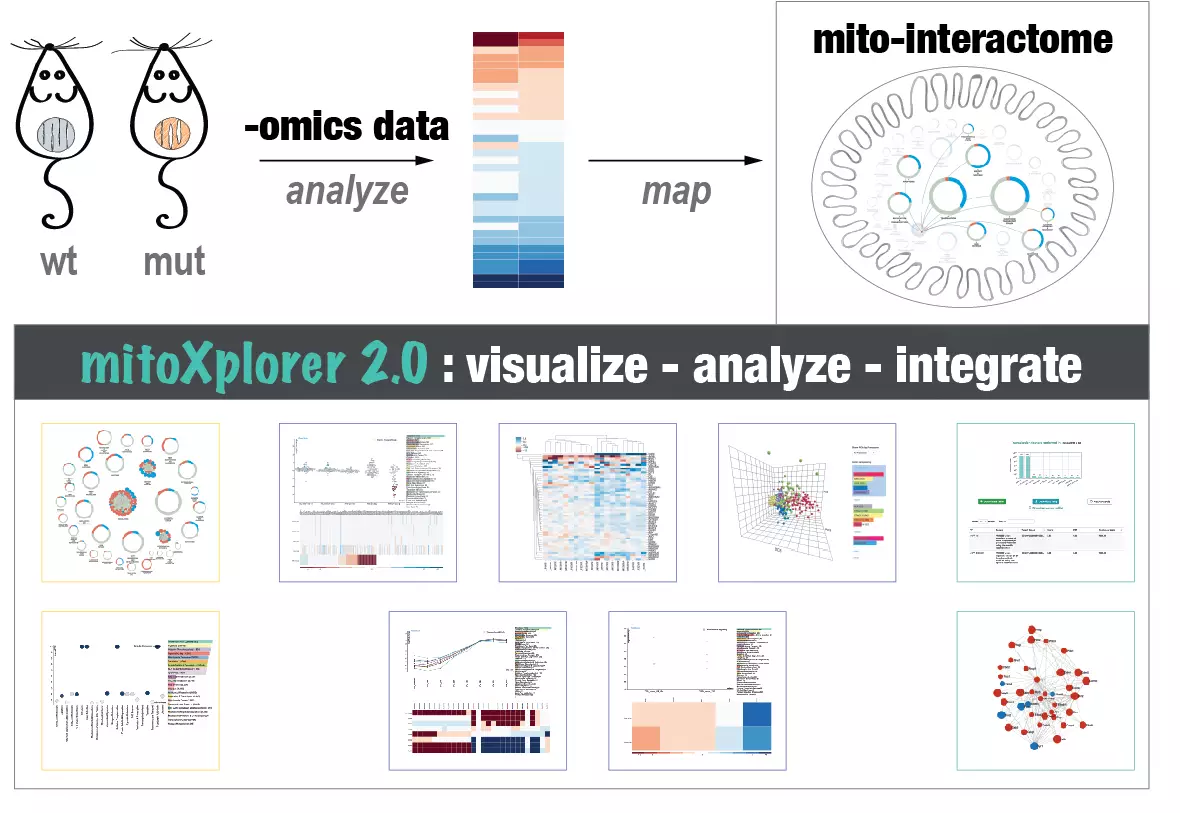
Biologie computationnelle mitochondriale : L’un de nos intérêts de recherche est de comprendre l’hétérogénéité mitochondriale et la diversité de la structure, de la fonction et de la dynamique d’expression à travers différents tissus, mais aussi dans différentes conditions de maladie. Nous utilisons des techniques de big data et d’intégration de big data pour apprendre comment les mitochondries s’adaptent à leur environnement cellulaire. Notre plateforme de visualisation et d’intégration de données mitoXplorer (http://mitoxplorer2.ibdm.univ-mrs.fr) nous aide à utiliser les données omiques pour étudier ce comportement mitochondrial.
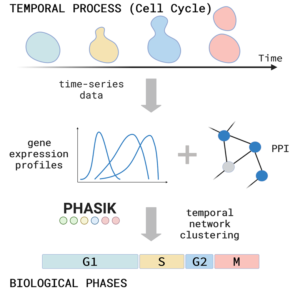
Biologie des réseaux: Dans un deuxième projet, nous utilisons des réseaux complexes pour étudier le comportement temporel des systèmes biologiques – surveillé par la dynamique temporelle de l’expression des protéines ou des ARN. A l’aide de ces techniques, nous pouvons également démêler les différentes phases de la dynamique d’expression mitochondriale dans le développement, le vieillissement ou dans la progression des maladies.
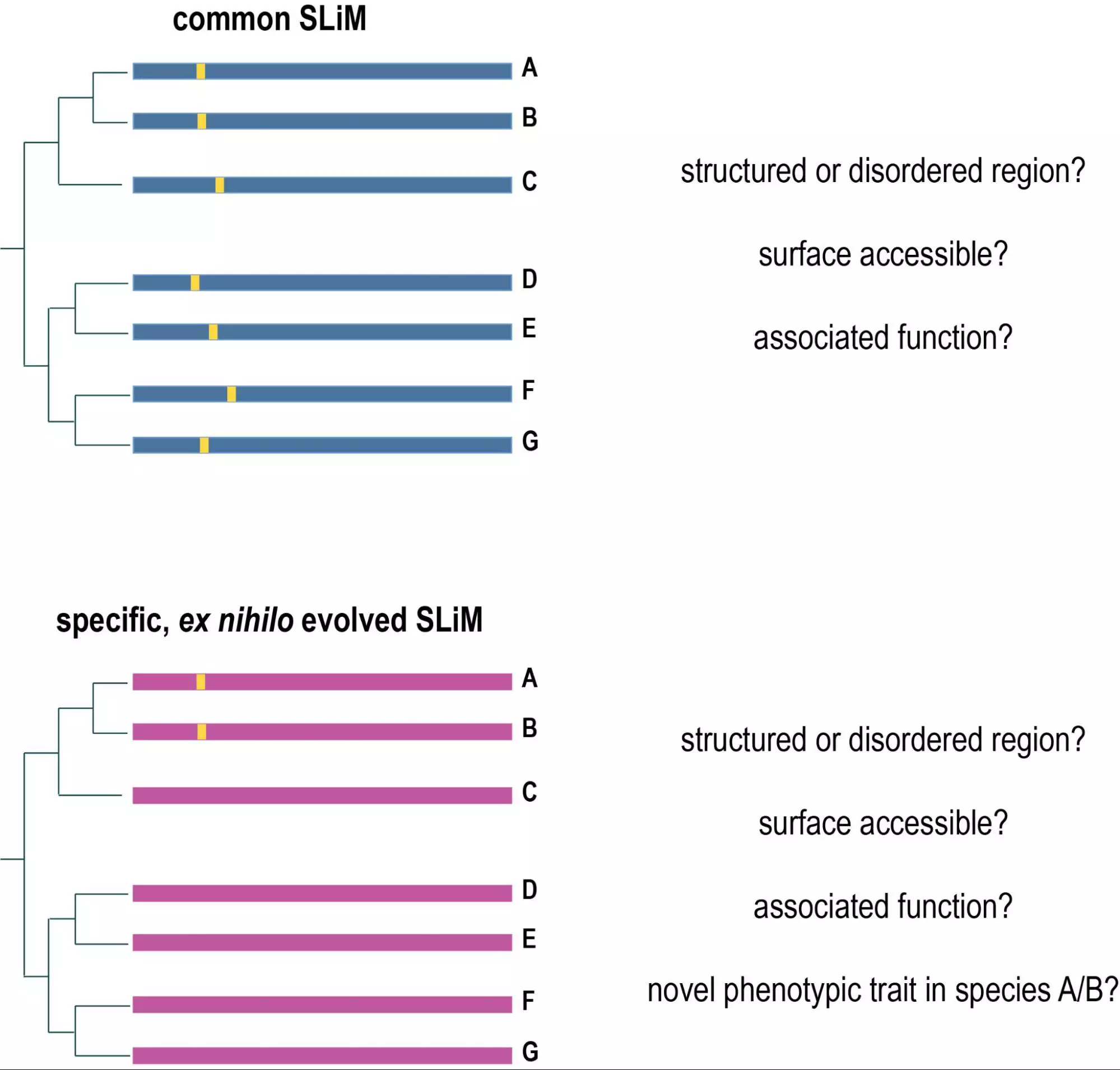
Biologie computationnelle évolutive:Nous nous intéressons également à la biologie computationnelle évolutive au niveau des séquences, des cellules et des organismes. Nous avons actuellement plusieurs projets liés à la biologie computationnelle évolutive. Au niveau des séquences, nous étudions l’évolution des motifs linéaires courts dans les protéines (SLiMs) et regardons spécifiquement les motifs prédateurs, ainsi que les motifs mécanosensibles (en collaboration avec l’équipe de Tam Mignot). A un niveau cellulaire, nous examinons l’évolution des épithéliums dans les organismes métazoaires les plus primitifs (éponges d’eau de mer) comme une structure importante pour définir les frontières de l’organisme et des tissus (en collaboration avec André le Bivic et Carole Borchiellini). Au niveau de l’organisme, nous nous intéressons à l’évolution des traits prédateurs chez les bactéries (en collaboration avec le laboratoire de Tam Mignot).
Développement d’outils informatiques pour l’exploration et l’intégration de données biologiques: Notre laboratoire développe des outils conviviaux pour l’analyse générale des données, la fouille de données et l’intégration de données pour la communauté des chercheurs. Découvrez ici ce que sont ces outils et où les trouver.
Publications
The mitoXplorer 2.0 update: integrating and interpreting mitochondrial expression dynamics within a cellular context
Evolution of mechanisms controlling epithelial morphogenesis across animals: new insights from dissociation-reaggregation experiments in the sponge Oscarella lobularis
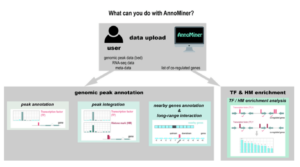
AnnoMiner is a new web-tool to integrate epigenetics, transcription factor occupancy and transcriptomics data to predict transcriptional regulators
RNfuzzyApp: an R shiny RNA-seq data analysis app for visualisation, differential expression analysis, time-series clustering and enrichment analysis
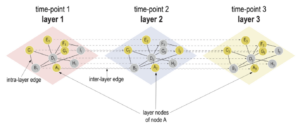
Introducing the novel Cytoscape app TimeNexus to analyze time-series data using temporal MultiLayer Networks (tMLNs)
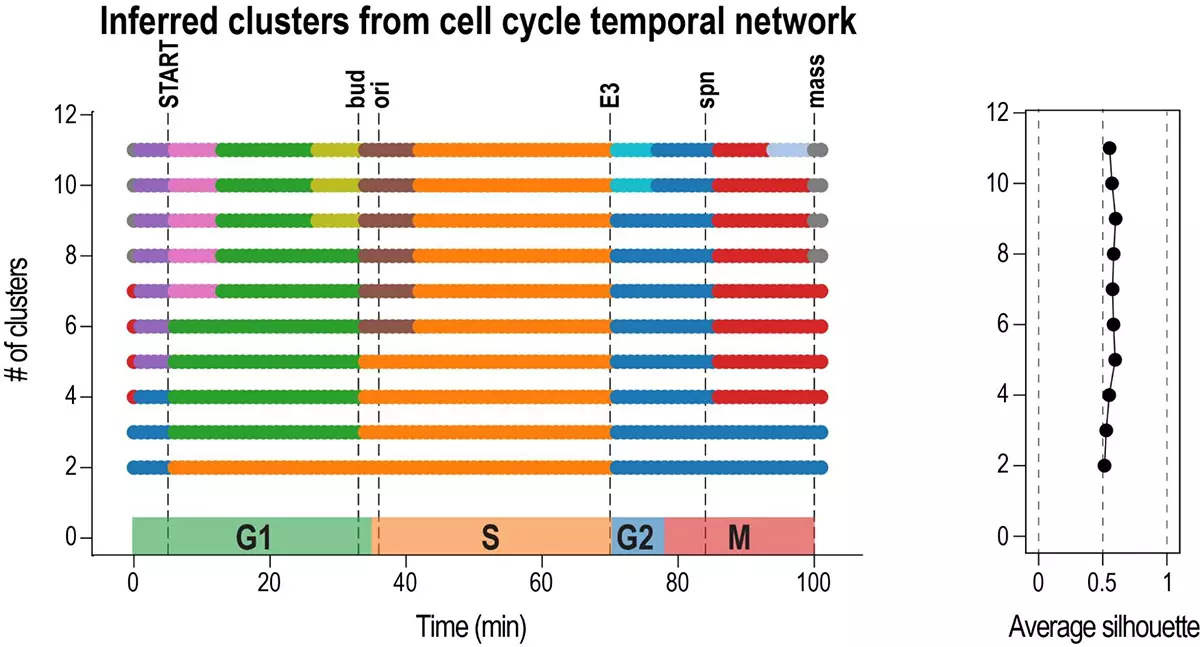
Inferring cell cycle phases from a partially temporal network of protein interactions
The mitoXplorer 2.0 update: integrating and interpreting mitochondrial expression dynamics within a cellular context
Evolution of mechanisms controlling epithelial morphogenesis across animals: new insights from dissociation-reaggregation experiments in the sponge Oscarella lobularis
AnnoMiner is a new web-tool to integrate epigenetics, transcription factor occupancy and transcriptomics data to predict transcriptional regulators
RNfuzzyApp: an R shiny RNA-seq data analysis app for visualisation, differential expression analysis, time-series clustering and enrichment analysis
Introducing the novel Cytoscape app TimeNexus to analyze time-series data using temporal MultiLayer Networks (tMLNs)
Evaluating the landscape of gene cooperativity with receptor tyrosine kinases in liver tumorigenesis using transposon-mediated mutagenesis
Phenotypic and genomic comparison of Photorhabdus luminescens subsp. laumondii TT01 and a widely used rifampicin-resistant Photorhabdus luminescens laboratory strain
Hypermethylation of gene body CpG islands predicts high dosage of functional oncogenes in liver cancer
A transcriptomics resource reveals a transcriptional transition during ordered sarcomere morphogenesis in flight muscle.
Integrative analysis and machine learning on cancer genomics data using the Cancer Systems Biology Database (CancerSysDB).
The deregulated microRNAome contributes to the cellular response to aneuploidy.
SLALOM, a flexible method for the identification and statistical analysis of overlapping continuous sequence elements in sequence- and time-series data
The axolotl genome and the evolution of key tissue formation regulators.
High-resolution TADs reveal DNA sequences underlying genome organization in flies.
HH-MOTiF: de novo detection of short linear motifs in proteins by Hidden Markov Model comparisons
Revision and reannotation of the Halomonas elongata DSM 2581T genome.
A Guide to Computational Methods for Predicting Mitochondrial Localization.
Oh Brother, Where Art Thou? Finding Orthologs in the Twilight and Midnight Zones of Sequence Similarity
Structure of a Cytoplasmic 11-Subunit RNA Exosome Complex.
Secretory cargo sorting by Ca2+-dependent Cab45 oligomerization at the trans-Golgi network.
Human Holliday junction resolvase GEN1 uses a chromodomain for efficient DNA recognition and cleavage.
Virtual pathway explorer (viPEr) and pathway enrichment analysis tool (PEANuT): creating and analyzing focus networks to identify cross-talk between molecules and pathways.
Tools for visualization and analysis of molecular networks, pathways, and -omics data.
The RNA-binding protein Arrest (Bruno) regulates alternative splicing to enable myofibril maturation in Drosophila flight muscle.
Merging and scoring molecular interactions utilising existing community standards: tools, use-cases and a case study.
morFeus: a web-based program to detect remotely conserved orthologs using symmetrical best hits and orthology network scoring.
PsicquicGraph, a BioJS component to visualize molecular interactions from PSICQUIC servers.
KEGGViewer, a BioJS component to visualize KEGG Pathways.
MTERF1 binds mtDNA to prevent transcriptional interference at the light-strand promoter but is dispensable for rRNA gene transcription regulation.
Designing efficient and specific endoribonuclease-prepared siRNAs.
HMMerThread: detecting remote, functional conserved domains in entire genomes by combining relaxed sequence-database searches with fold recognition.
SeLOX–a locus of recombination site search tool for the detection and directed evolution of site-specific recombination systems.
Genome-wide resources of endoribonuclease-prepared short interfering RNAs for specific loss-of-function studies.
ProFAT: a web-based tool for the functional annotation of protein sequences.
An Ambystoma mexicanum EST sequencing project: analysis of 17,352 expressed sequence tags from embryonic and regenerating blastema cDNA libraries.
DEQOR: a web-based tool for the design and quality control of siRNAs.
The BAR-domain family of proteins: a case of bending and binding?
The power and the limitations of cross-species protein identification by mass spectrometry-driven sequence similarity searches.
Actualités
Félicitation à Robert Kelly, Frank Schnorrer, Cédric Maurange, Bianca Habermann et Delphine Delacour !
L’IBDM inspire les jeunes esprits : en impliquant les enfants des écoles primaires dans la lutte contre le cancer pédiatrique (“Contre le cancer, j’apporte ma pierre”) et en interagissant avec les lycéens grâce à des expériences immersives (DECLICS).
Rejoignez-nous le 29/06/2023 à 12:30 dans l’Amphi 12 pour une présentation passionnante deux des membres de notre équipe : Rikesh Jain et Theo Brunet !
Montre-moi ton rythme !
Phasik est un algorithme permettant d’extraire les phases des systèmes biologiques en regroupant des réseaux temporels partiels.
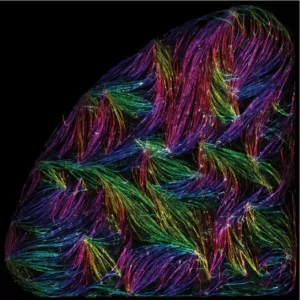
Les cellules musculaires s’auto-organisent en faisceaux de fibres in vitro, sans la présence de signaux externes !
Nous présentons ‘AnnoMiner’, un nouvel outil convivial basé sur le web pour annoter et intégrer les données épigénétiques et de liaison des facteurs de transcription.
L’équipe Habermann a adapté le concept des réseaux multicouches généralement utilisés pour intégrer différents types de données.
Notre équipe à l’IBDM participe à un concours de maître de conférence pour un poste de professeur assistant permanent en bio-informatique à Aix-Marseille Université.
Membres de l'équipe
Alumni
Les organismes qui nous financent